英文名:CrY2H-seq: a massively multiplexed assay for deep-coverage interactome mapping
发表单位:美国加利福尼亚Salk生物研究所遗传分析实验室
杂志:Nature Methods,2017
影响因子:25.062
阅读人:罗辑
摘要:
大规模的蛋白互作网络图谱绘制是一项很大的挑战,因为涉及成本,时间,现有技术的灵敏度限制等。这篇文章介绍了一种大规模、多复用的酵母双杂交方法。CrY2H-seq是将Cre重组酶报告基因在细胞内融合互作蛋白的编码序列,以及使用二代测序方法来全面鉴定这些互作网络。我们应用CrY2H-seq方法来研究稀少的有注释的拟南芥转录因子的相互作用。通过十组独立的筛分实验,测试了3600万组互作组合,覆盖了1453个转录因子的8577个互作反应网络,我们证明了CrY2H-seq方法更加优良的筛选能力,效率和比现有技术更强的灵敏度。我们把这种深度覆盖网络资源称为AtTFININ-1,其概括了1/3以往报道的根据不同方法获得的相互作用信息,扩展了三倍已知植物转录因子相互作用的数量,揭示了一些未知的家族特异性互作模块,这些模块与植物的生殖发育,根系骨架和生理节律相关。
研究背景:
酵母双杂交实验是目前最为广泛使用的绘制蛋白互作的方法之一。此实验的数据有助于丰富蛋白互作的资源库,还可以帮助科研工作者鉴定癌症亚型和可能的与植物病原菌感染相关的子网络。但是大规模的酵母双杂交实验数据采集和重复筛选受到成本和劳动力资源限制。而利用二代测序手段来鉴定蛋白互作的方法使得酵母双杂交筛选变为可行方案。起初科研工作者使用的是条码融合遗传方法(BFG-Y2H),其原理是利用条形码的开放阅读框克隆区域的细胞内DNA重组来鉴定互作蛋白,从而允许Y2H阳性克隆被同时合并及测序。但是这种技术由于需要对每个条码诱饵分子和捕获分子分离和测序,所以价格十分昂贵。为了更加高效的进行重复筛选,我们开发了Cre报告基因间接调解酵母双杂交,并结合二代测序的方法(CreY2H-seq)。此方法是利用Cre重组酶作为Y2H蛋白互作报告因子,在细胞内通过特异识别蛋白编码序列的loxP位点,共价单向的连接互作的诱饵和捕获质粒。连接的蛋白编码序列充当互作识别DNA分子,结合二代测序手段,能够大规模多重复的筛选,监测蛋白互作网络。我们使用CreY2H-seq技术全面筛选了1956个拟南芥转录因子和调控因子。从十组独立筛选实验中,我们报道了8577个转录因子互作组,其中7994个互作组是之前从未报道过的。在经过实验和计算方法的验证后,我们鉴定了几个与植物的生殖发育,根系骨架,生理节律的环境调控,胁迫和激素反应通路干扰相关的网络模块。
研究目的
应用CrY2H-seq方法来研究稀少的有注释的拟南芥转录因子相互作用。通过十组独立的筛分实验,测试了3600万个互作组,覆盖了1453个转录因子的8577个反应网络,证明了CrY2H-seq方法更加优良的筛选能力,效率和比现有技术更强的灵敏度。
材料方法
1. 实验材料
通过基因克隆表达构建了1956个转录因子文库。
2. 分析方法
1956个转录因子分别进行CrY2H-seq方法,二代测序方法,HT-Y2H方法,wNAPPA方法。
研究结果
1. CrY2H-seq方法的开发:首先培养酵母菌株CRY8930, 其携带了Gal4诱导的GAL7::CRE表达盒和两个典型的营养缺陷型的表达盒(GAL1::HIS3和 GAL2::ADE2)(Fig. 1a)。我们优化了ARS/CEN 通路兼容质粒,包括单向脂氧合酶序列,从而使得所有ORF插件在固定位置都是相同的DNA分子(Fig. 1b)。通过筛选质粒中酵母转化子的已知阳性和阴性互作对,我们确定了阳性互作对引导了Cre表达和选择生长。酵母菌落PCR与Gal4-AD和Gal4-DB引物(Fig. 1b)产生阳性互作组的扩增子,表明质粒经过Cre重组。Cre重组PCR产物的Sanger序列法证实一种新形式的双重突变脂氧合酶位点。这个位点像三明治一样,夹在两个ORF序列中间,以一种3′-end-to-3′-end形式进行重组(Fig. 1c)。并且不管使用CRY8930或未优化的Y8930,都能给出阳性和阴性的相互作用结果。
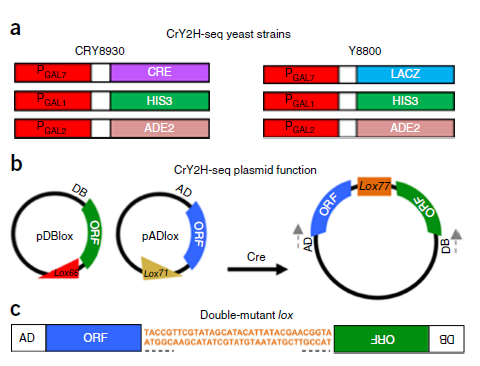
图1. CrY2H-seq菌株和质粒设计
CrY2H-seq技术与目前的多复用Y2H技术有两个明显的区别。第一,由CrY2H-seq方法检测的相互作用需要两个报告基因的平行激活;我们使用HIS3结合CRE是因为GAL1:HIS3能比ADE2更为敏感地检测互作蛋白,并且使用独立的GAL7启动子驱动CRE表达可降低启动子特异性的假阳性结果。并且包括CRE作为次级报告基因最小化了甾类化合物诱导的Cre表达系统所需要的时间和试剂。第二,CrY2H-seq技术利用互作蛋白编码序列自身来形成胞内的DNA识别子(Fig. 1c),而不是会成为大规模筛选瓶颈障碍的条码识别子。这些关键特征让我们可以规避现有的Y2H局限性,并且可以建立一个通用的CrY2H-seq流程来进行大规模多复用的筛选(Fig. 2)。
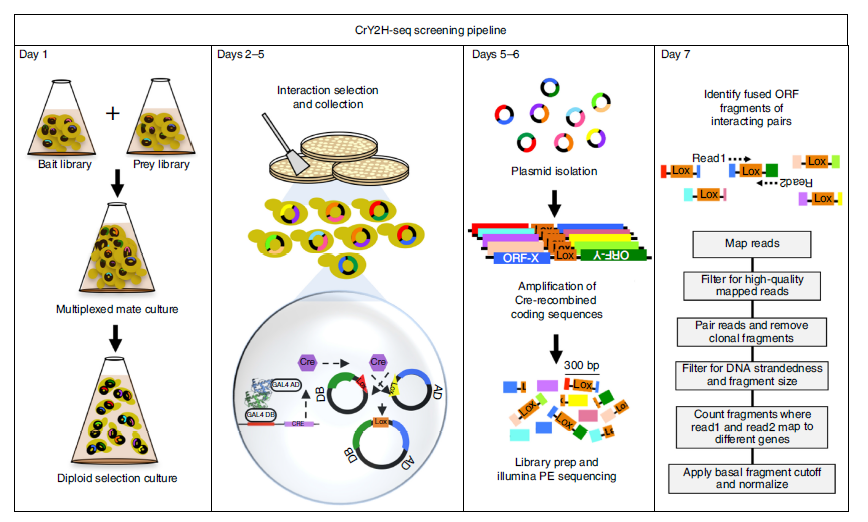
图2. CrY2H-seq筛选流程图
2. 拟南芥转录因子ORF组的深度互作筛选:我们载入1956个拟南芥TFs到CrY2H-seq流程中,对最终的诱饵和捕获库,分别包括1877和1933个独特的酵母克隆进行了十组全面筛选。初始文库显示了与预期一致的ORF粒径分布,数据也显示了最小的ORF大小误差。虽然在进行Y2H筛选之前,屏蔽了诱饵蛋白的自我激活,我们还是选择剔除了这个步骤,目的是想挑战尝试这种实验方法是否监测的是真实的互作反应信号,而不是自我激活互作反应的噪音信号。为了控制自我激活的诱饵蛋白,我们对每组筛选加入超量的含有空pADlox质粒的Y8800菌株。然后文库配对及重复十次HIS3报告基因选择。深度筛选检测了3600万潜在的蛋白组差不多300次,共约10亿组反应。
对每组筛选进行了质粒的多模板PCR扩增后,我们随机修剪PCR产物至大约300bp,生成标准的以Illumina为基础的DNA序列文库(Fig. 2)。我们进行了100bp测序,旨在预先建立优化后的覆盖范围(4000万个读数点/每组筛选)。对测序读数进行绘制和质量筛选,与Cre重组ORF一致的片段被提取出来。我们应用了一个基准片段,来排除少于三个连接片段代表的互作分子。其他互作鉴定片段的十次筛选结果经过标准化处理,以控制测序数据之间的误差,他们被归类为‘标准化的蛋白互作片段’(NPIFs; Fig. 2)。最小限度的扩增子粒径误差在我们的数据集中也被发现,虽然二聚体的片段绘制没有出现在数据中,有可能是因为融合ORFs的夹发针结构很难扩增,这在之前的小规模筛选实验中也被发现了。
共计1.0900亿NPIFs被鉴定,这些NPIFs对应的是173000个独特的Cre重组ORF连接组(Fig. 3a)。其中299个不同的pDBlox ORFs融合到空的pADlox载体中,表明16%的诱饵蛋白显示了自我激活能力。164293个独特的包含这些TFs的ORF组从数据中排除。余下的140万(13%)的NPIFs对应了8577个蛋白相互作用,即每个反应里平均有7个NPIFs(Fig. 3b)。最终8577组相互作用反应构成了拟南芥转录因子互作网络。
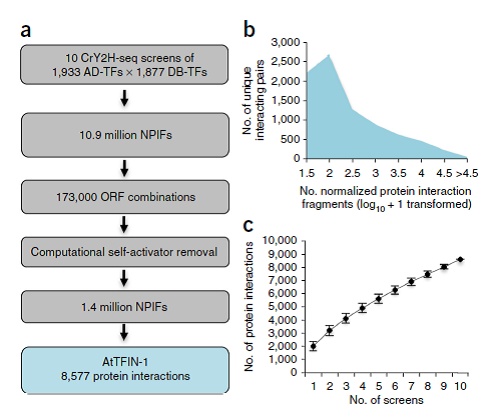
图3. AtTFIN-1覆盖范围
3. AtTFIN-1互作反应的确认:为了评价样品灵敏度,每组筛选中所有可鉴定的互作反应部分,我们对重复筛选的所有可能排序都模拟了结果,发现每一个筛选产生了平均2,012 ± 354 组相互作用。计算十次筛选后得到的新的相互作用平均值,发现并没有达到饱和。我们通过Michaelis–Menton 模型拟合曲线来判断饱和度和确定了15,610 ± 2,661 组反应能被检测,超过了一半的数量(54.6%)。
为了评价重复性,我们复检了771(9%)组AtTFIN-1反应(其中678组是新的互作反应)。除了自我激活的诱饵蛋白外,我们观察到有73%(422/580组新的互作反应和57/76组已知互作反应)被再次检测到。另外,我们发现在并联筛选中出现了更高的复检率,而在不同范围的NPIFs的相关互作反应里却呈现相似的复检率。我们也用wNAPPA方法检测了94组AtTFIN-1反应(59组是新的反应组),发现50%的反应组和25.4%的新反应组是阳性结果(Fig. 4b)。
为了评价实验灵敏度,所有可检测的生物物理互作反应部分,我们挖掘了CrY2H-seq 技术里检测到的TF反应所涉及的文献报道和数据库。我们把这些从文献或者数据库挖掘出来的反应统称为已知互作反应。涉及自我激活TFs的反应和二聚体都被排除在外。AtTFIN-1显示了与拟南芥互作组-1反应有着领先的重叠率(52.2%),而与AraNet互作反应有着最小的重叠率(13.4%)(Fig. 4c)。我们通过计算十次数据的AtTFIN-1反应重叠率,判断假阳性大概占到0.69% ± 0.12%。综上,相对于随机互作,AtTFIN-1相互作用显示了已知相互作用的更强重复性(Fig. 4c)。所有检测率的精度回归曲线显示在一次筛选和两次筛选之间在精度上有很大的差别,指引我们在两次或更多次的筛选结果中归类高可信度的相互作用。
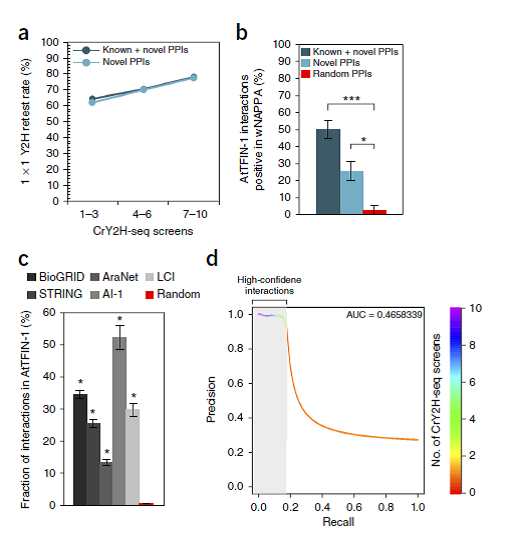
图4. AtTFIN-1的质量
为了测试其优化后性能优于高通量Y2H(HT-Y2H),针对拟南芥互作组-1,我们比较了分别用CrY2H-seq 和HT-Y2H方法测试的TF相互作用的检测率。CrY2H-seq显示了比HT-Y2H相比,高达5倍的检测率增长。对于普通的筛选过的TF 相互作用,CrY2H-seq方法显示了7倍的检测率增长,覆盖了1609组TF相互作用,而HT-Y2H只检测到229组互作。对于普通的文献策划的互作组,CrY2H-seq重现了33.3%的检测率,而HT-Y2H只显示12.3%。尽管CrY2H-seq显示了比HT-Y2H明显的改进,但是应当指出的是拟南芥互作组-1是基于两次主要的筛选,而且经过成对的二次测试筛选,而AtTFIN-1是基于十次主要筛选,并且没有经过二次检测筛选。
为了评价AtTFIN-1相互作用的生物相关性,我们比较了AtTFIN-1互作组和6057个不同表达数据集的随机互作数据进行表达相关性的比较。我们发现了AtTFIN-1在转录编码互作里有更显著的表达相关性,这一发现支持了AtTFIN-1体内相互作用的潜力。
4. AtTFIN-1定义扩展的转录因子模块:我们通过寻找经常出现的这种‘优先的’家族间和家族内相互作用,进一步研究了3086个高可信度的AtTFIN-1相互作用(2578组新互作)的生物显著性。AtTFIN-1相互作用通过以往指定的家族进行分类,然后与10000个随机保守的互作网络进行比较(Fig. 5a)。我们在已知的可二聚化的家族成员中发现了显著的优先家族间和家族内相互作用,这些家族包括bHLH, MADS, bZIP, NAC, WRKY, AUX-IAAs和ARF。我们也在植物特定家族间发现了显著的优先家族件和家族内相互作用,包括生长调控因子(GRFs)和生长调控因子互作因子(GIFs),LUGs和YABBYs,及AUX-IAAs和ARFs。类蜀黍分支/环形棘皮纲/增殖细胞因子(TCP)家族对18个TF家族来说表现了显著的优先性,这种广泛的优先性是与TCPs核心蛋白以往的研究数据是一致的。
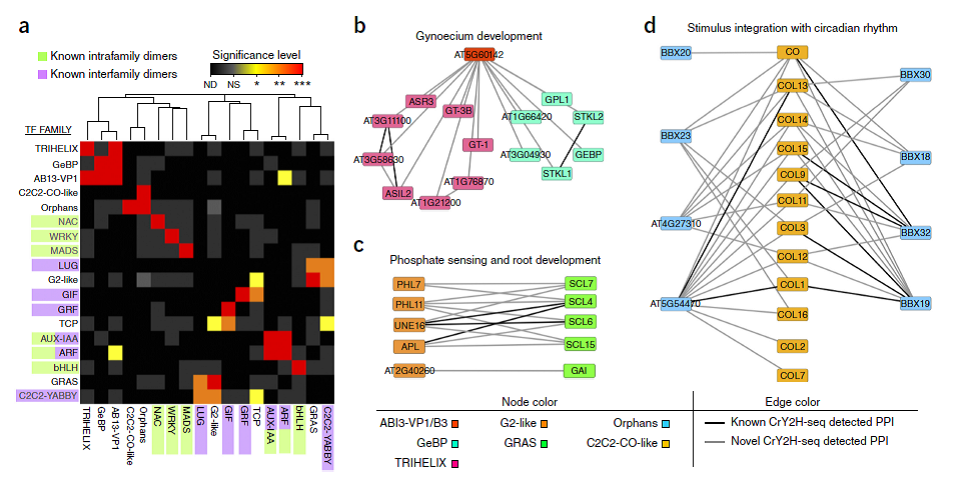
图5. AtTFIN-1中涵盖TF家族互作的生物功能
我们进一步测试了高显著的,以往未报道的优先家族内相互作用;发现在ABI3-VP1/B3 家族成员AT5G60142的驱动下,ABI3-VP1/B3 家族对GeBP和TRIHELIX 蛋白的优先性,即有很多与各种TRIHELIX 和 GeBP 成员之间的相互作用(Fig. 5b)。尽管TRIHELIX 和 GeBP成员只有很少的GO注释,但AT5G60142已在分离的早期雌蕊内侧区域的细胞内被发现上调。有趣的是,不仅是AT5G60142,93%(13/14)的TRIHELIX 和 GeBP互作对都在这个研究里面被发现共表达现象,但是5个AT5G60142互作对(ASIL2, AT3G58630, AT1G76870, AT3G04930和STKL1)在细胞内同一区域被发现显著上调。这些互作可能形成部分以往未被识别的与早期生殖发育相关的模块。我们也发现在多重的phosphate-response-like因子和scarecrow-like因子的驱动下, G2-like蛋白对GRAS家族的优先性(Fig. 5c)。这个网络揭示了磷酸盐传感和根系发育的逻辑联系,与磷酸盐缺失导致改变根系构型和增加根须密度的概念是一致的。C2C2-CO-like TFs表明与未定义的TFs孤一种类有着显著的优先相互作用(Fig. 5d)。这些互作反应的进一步检测揭示了所有含有BBX结构域的蛋白,包括C2C2-CO-like蛋白自身。这些相互作用可以被BBX调解,因为这些结构域已经被报道在蛋白互作调解和转录调控中起着很重要的作用。许多含有BBX结构域的蛋白都有特异的,有时候甚至是相反的功能,作用于调控花朵,生物钟,生物的和非生物的胁迫反应。而且,目前报道已指出在大豆植物里通过改变光照强度和不同生殖发育阶段的时钟基因的表达模式,从而过表达AtBBX32,增加粮食产量。AtTFIN-1模块认为BBX蛋白的组合复杂性有可能通过反馈或前馈环路等方式在整合环境信号和开花时间方面发挥很重要的作用。
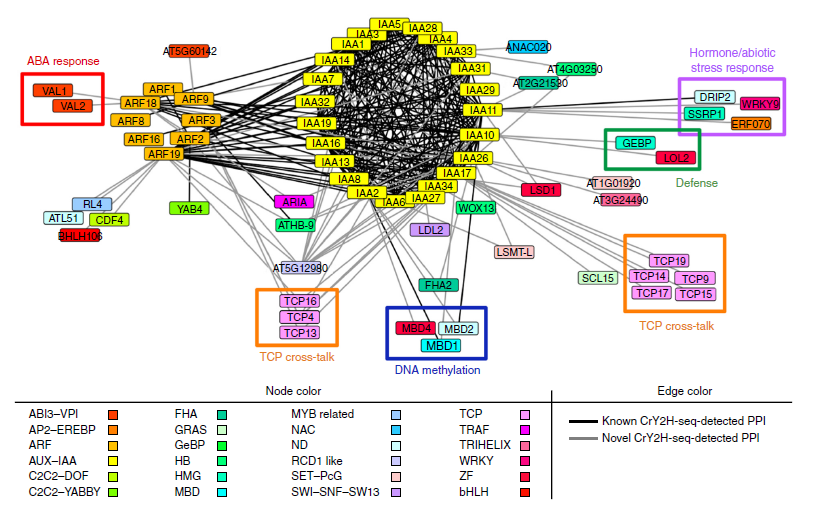
图6. 扩展的ARF–AUX–IAA转录因子网络
除了ARFs和AUX/IAAs之间的家族内相互作用外,个别的AUX-IAA成员显示与其他家族也有独特的相互作用(Fig. 6)。例如,与其他IAAs相比,IAA17与TCPs有很强的相互作用;这表明IAA17可能在生长素和TCP转录调控之间起着重要的调解作用。IAA2, IAA10, IAA17和IAA18通常与甲基-CpG结合域(MBD)蛋白相互结合,表明这些IAAs在调控DNA甲基化方面有着潜在的牵连。独特的IAAs和ARFs表明与特异的激素有相互作用,胁迫相关的TFs-IAA11与激素或非生物的胁迫响应因子ERF70和DRIP2有结合,IAA10和防御响应因子LOL2和GEBP有相互作用,ARF18和脱落酸响应因子VAL1和VAL2。这表明这些因子在整合生长素响应和不同激素和胁迫信号方面的巨大潜能。扩展的ARF–AUX–IAA互作组揭示了TFs在调解生长素响应和其他植物通道的相互作用方面如何发挥其独特的作用。
创新点及启示:
1、 利用一种新技术,把酵母双杂交和二代测序方法相结合,可以大规模、多复用的深度覆盖互作网路绘图
CrY2H-seq:一种研究蛋白与蛋白相互作用的手段,名为蛋白质互作网络绘制手段。
原理如下:
首先将编码蛋白质的基因分别连在两个质粒上再导入细胞;如果两种蛋白在细胞内发生互作,质粒上的Cre基因就会被激活,在它的牵引下两个独立质粒上的两种基因就会连在一起。通过 Cre重组酶在细胞内把两个相互作用蛋白的编码基因连接在一起,然后通过下一代DNA测序技术大规模地鉴定这些可以互相作用的蛋白基因。
抗体组优势:
1. 抗体组技术对拟南芥转录因子蛋白互作网络进行深度研究以后,可以直接与拟南芥的转录组和基因组数据库进行相关性联合分析,可以冲击更高影响因子的文章;
2. 从研究深度来说,文章中提到的CrY2H-seq技术,是通过基因测序数据推算出相互作用的蛋白分子;而抗体组技术可以直接锚定任何蛋白分子之间的相互作用,所以对客户来讲,抗体组技术研究蛋白互作网络更加直接高效;
3. 从灵敏度角度来讲,对于转录因子这种低丰度蛋白,抗体组技术可以更灵敏的捕捉和提取,得到的蛋白互作网络信息可能更加全面和丰富,客户有可能通过发现的新蛋白或者通路数据来冲击更高影响因子的文章;
4. 从覆盖度角度来说,抗体组技术也同样可达到文章提到的互作组规模,进行高通量大规模的筛选;
艾比玛特医药科技(上海)有限公司
上海市徐汇区桂平路333号聚科生物园区1号楼1-3层
邮箱:market@ab-mart.com
应聘职位:hr@ab-mart.com
订购专线:4006-123-828
销售电话:13916964679(微信同号)
技术支持:15618194176(微信同号)
华南经销商负责(广东,广西,福建,海南):
程经理:手机18616261485(微信同号)
华北经销商负责(北京,天津,河北):
徐经理:手机15618191473(微信同号)
南方经销商负责:
陆经理:手机13122837132(微信同号)
北方及西南经销商负责:
张经理:手机13122150513(微信同号)

微信客服
邮箱:market@ab-mart.com
应聘职位:hr@ab-mart.com
订购专线:4006-123-828
销售电话:13916964679(微信同号)
技术支持:15618194176(微信同号)
华南经销商负责(广东,广西,福建,海南):
程经理:手机18616261485(微信同号)
华北经销商负责(北京,天津,河北):
徐经理:手机15618191473(微信同号)
南方经销商负责:
陆经理:手机13122837132(微信同号)
北方及西南经销商负责:
张经理:手机13122150513(微信同号)

微信客服
沪ICP备17056956号-2 艾比玛特医药科技(上海)有限公司